[项目学习]PyHealth-临床预测建模的深度学习工具箱
开箱即用,集成了一部分模型(CNN、LSTM、GRU…),支持多个开源数据(MIMIC3 MIMIC4 eICU…),也支持个人数据集(提供了函数将样本处理成json格式的数据,在这个json数据中,有一个标签label,通过这个标签去控制这个任务是什么类型的任务,二分类、多分类…),可支持“基于诊断的药物推荐”、“患者住院和死亡率预测”、“ICU停留时间预测”等医疗任务。与任务独立,支持多个数据
会议/期刊:KDD’23 Tutorial
作者: Chaoqi Yang, Zhenbang Wu, Patrick Jiang, Zhen Lin, Junyi Gao, Benjamin P. Danek,…
机构: UIUC, UE
主页: PyHealth
代码: Github
教程: google drive
包含任务类型:
- 临床预测建模:readmissiong prediction, length of stay prediction task,mortality prediction, diagnosis-based drug recommendation
- 生理信号的深度学习:sleep staging, EEG event detection, abnormal EEG detection, arrythmia detection
- 医学图像:x-ray representation learning, chest disease classification, medical report generation
- 生物医学文本:clinical notes classification and medical report generation
- 预训练模型相关:Medical concept lookup, mapping and embeddings
PyHealth简介
『说明』:开箱即用,集成了一部分模型(CNN、LSTM、GRU…),支持多个开源数据(MIMIC3 MIMIC4 eICU…),也支持个人数据集(提供了函数将样本处理成json格式的数据,在这个json数据中,有一个标签label,通过这个标签去控制这个任务是什么类型的任务,二分类、多分类…),可支持“基于诊断的药物推荐”、“患者住院和死亡率预测”、“ICU停留时间预测”等医疗任务。
医疗任务构建流程

- 安装(pip install pyhealth或git clone下在pyhealth目录下 pip install .)
- 这个包有三个关键功能,数据处理、多个模块的单独调用、医疗代码的映射
- 加载数据集(pyhealth.datasets)
- 定义任务函数(pyhealth.tasks)
- 构建ML/DL模型(pyhealth.models)
- 只要传入数据集、特征列、标签,然后告诉是什么类型(分类、回归…)任务即可
- 模型训练(pyhealth.trainer)
- 推理及评价(pyhealth.metrics)
模块介绍
pyhealth.datasets
与任务独立,支持多个数据集,输出的是一个多级字典结构,如:mimic3

以图的形式对患者的诊疗数据分层(逐级):字典
- 患者level,在这一层中包含患者的访问记录,人口统计学信息…,会有Visit属性
- 访问level(医院)
- 涉及事件:诊断、程序、开药(临床时序数据)
以图的形式将数据处理结果展示,最上层是不同患者,在这一层中除了包含第二层的Visit属性,还会包含患者本身信息,比如患者ID,visit_id(等价inhos_times),患者人口统计学信息,在第二层中细化了第一层的VISIT属性,同时在第二层又包含了第三层的事件列表属性,第二层中包含了患者在不同医院的就诊记录,第三层细化了第二层的事件列表,如某医院的就诊事件:诊断、用药…

pyhealth.tasks
定义如何将每个患者的数据处理为任务的一组样本。在这个包里,提供了几个任务示例,比如:drug recommendation和length of stay prediction
pyhealth.models
提供参数配置相似但模型不同的机器学习模型
pyhealth.trainer
指定训练参数,例如epoch、优化器、学习率等,训练器会自动保存最佳模型和最终输出路径
pyhealth.metrics
提供了几种常见的评估指标,对二分类、多分类或多标签分类设置与sklearn.metrics中的评价指标有相同的样式和参数,同时还对不同的任务,提供了模型校准的相关指标以及预测集评估的相关指标,此外还提供了不确定性量化的指标,对医疗任务也提供了专门的评价指标,比如:药物相互作用率(DDI)。详情见模型评价指标官方文档
Medical Code Map
- 函数:pyhealth.codemap,该函数提供了两个核心功能,可独立使用
- 功能1:用于在一个医学编码系统内进行代码本体查找(例如名称、类别、子概念)
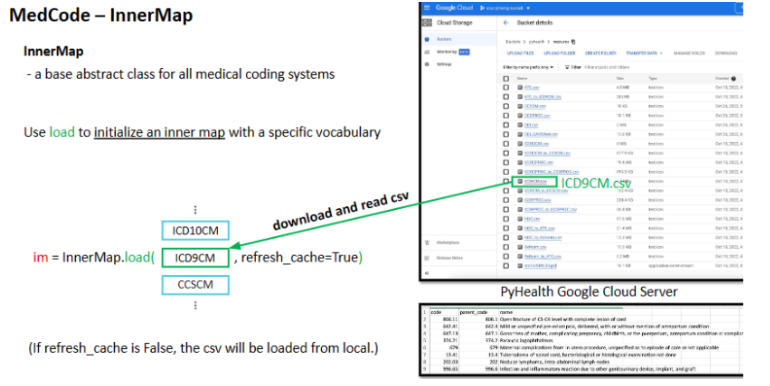
from pyhealth.medcode import InnerMap
icd9cm = InnerMap.load("ICD9CM") #提供了icd9
icd9cm.lookup("428.0")
# `Congestive heart failure, unspecified`
icd9cm.get_ancestors("428.0")
# ['428', '420-429.99', '390-459.99', '001-999.99']
atc = InnerMap.load("ATC")
atc.lookup("M01AE51")
# `ibuprofen, combinations`
atc.lookup("M01AE51", "drugbank_id")
# `DB01050`
atc.lookup("M01AE51", "description")
# Ibuprofen is a non-steroidal anti-inflammatory drug (NSAID) derived ...
atc.lookup("M01AE51", "indication")
# Ibuprofen is the most commonly used and prescribed NSAID. It is very common over the ...
- 功能2:两个编码系统之间的代码映射(比如:ICD9CM到CCSCM)
from pyhealth.medcode import CrossMap
codemap = CrossMap.load("ICD9CM", "CCSCM")
codemap.map("428.0")
# ['108']
codemap = CrossMap.load("NDC", "RxNorm")
codemap.map("50580049698")
# ['209387']
codemap = CrossMap.load("NDC", "ATC")
codemap.map("50090539100")
# ['A10AC04', 'A10AD04', 'A10AB04']
一张图总结
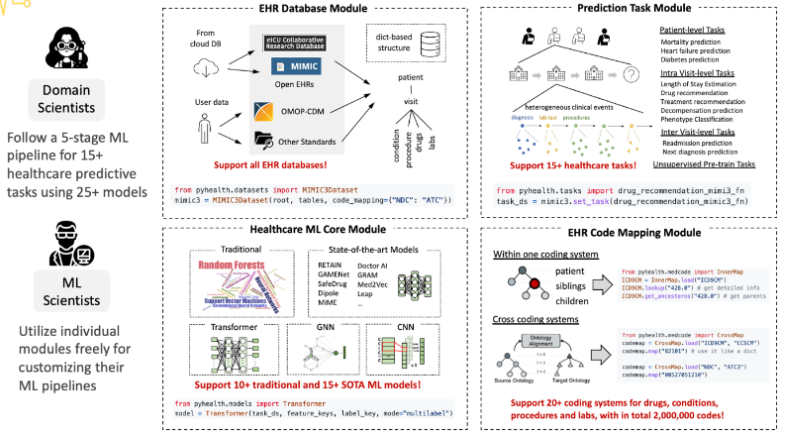
使用EHR进行临床预测建模
- EHR:Electronic Health Records(电子健康记录)
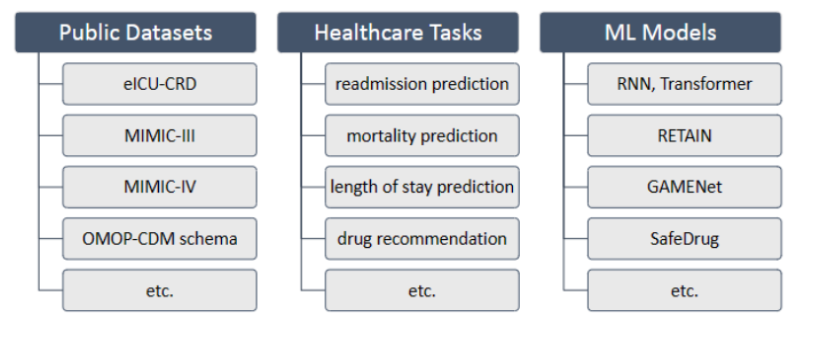
- 支持的EHR数据集:eICU-CRD、MIMIC数据集…
- 医疗任务:再入院预测、死亡率预测、停留时间预测、基于诊断的药物推荐等
- 机器学习模型:RNN、Transformer、RETAIN等
- 作者使用RETAIN模型在MIMIC-III数据集上做了停留时间预测任务的学习。代码链接
2016 NeurlPS:RETAIN:An Interpretable Predictive Model for Healthcare using Reverse Time Attention Mechanism
title:RETAIN模型简介
- RETAIN(REverse Time AttentIoN)模型是为患者死亡率预测任务提出的机器学习模型。该模型利用循环神经网络和注意力机制的组合来从电子健康记录 (EHR) 数据中学习。 RETAIN 模型将患者的纵向 EHR 数据作为输入,包括随时间推移的诊断、药物治疗和手术。然后,该模型根据患者的 EHR 数据生成死亡率预测评分。
- RETAIN 模型的独特之处在于它能够提供可解释的预测。模型中纳入的注意力机制使我们能够识别 EHR 数据中有助于预测的重要特征。这在医疗领域特别有用,因为可解释性对于患者护理和决策至关重要。
- RETAIN 模型已被证明可以有效预测重症监护病房患者的死亡率,并且性能优于其他机器学习模型。该模型在其他临床预测任务中具有潜在的应用,例如再入院预测和疾病进展预测。
生物信号的深度学习
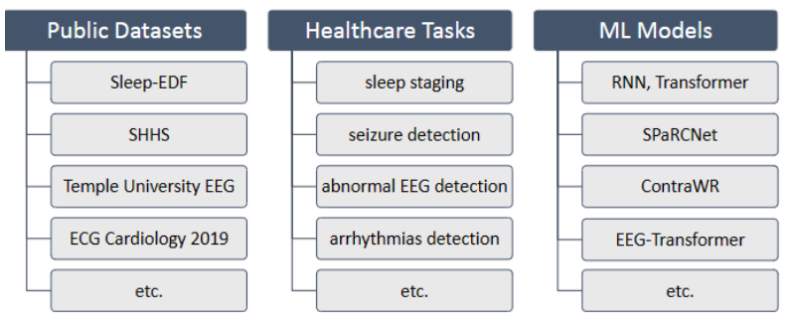
- 支持生物信号数据集:ISRUC、Cardiology、Sleep-EDF、SHHS、TUEV、TUAB等
- 支持的生物信号任务:睡眠分期、脑电时间检测、异常脑电波检测、心律失常检测等
- 支持的EHR模型: ContraWR、SPaRCNet、ST-Transformer 等
- 作者使用ContraWR模型在Sleep-EDF数据集上执行了睡眠分期任务:预测30s信号的睡眠阶段标签 代码路径
2023JMIR:Self-supervised Electroencephalogram Representation Learning for Automatic Sleep Staging
title:ContraWR简介
- ContraWR(Contrastive Word Repetition)模型是一种用于文本分类的深度学习模型。该模型使用了反向重复学习的思想,首先将输入的文本进行石膏化处理,然后结合双向LSTM模型捕获文本的上下文信息。接着通过多层注意力机制对不同层次的上下文信息进行加权,最后利用Softmax分类器对文本进行分类。
- ContraWR模型采用了对抗训练策略,通过随机替换原始文本中的短语来生成虚假的训练样本,这样可以提高模型对于噪声的鲁棒性。此外,ContraWR还引入了构造对抗样本的技术,以增强模型的鉴别能力。
- ContraWR模型在多个文本分类任务上表现出很好的性能,比如情感分类、垃圾邮件过滤、新闻分类等。除此之外,通过对抗学习策略的应用,ContraWR模型还对一些文本攻击具有强的抵御力,这种模型可以在各种应用场景中广泛应用。
医学影像分析
- 支持的医学图像数据集:CheXpert、RSNA、COVID和MIMIC-CXR
- 支持的任务:X射线表示学习、胸部疾病分类、医疗报告生成
- 支持的模型:RNN、ResNet…(可能有关神经网络的模型都可以用)
- 使用ResNet在COVID数据集上演示胸部疾病分类演示代码
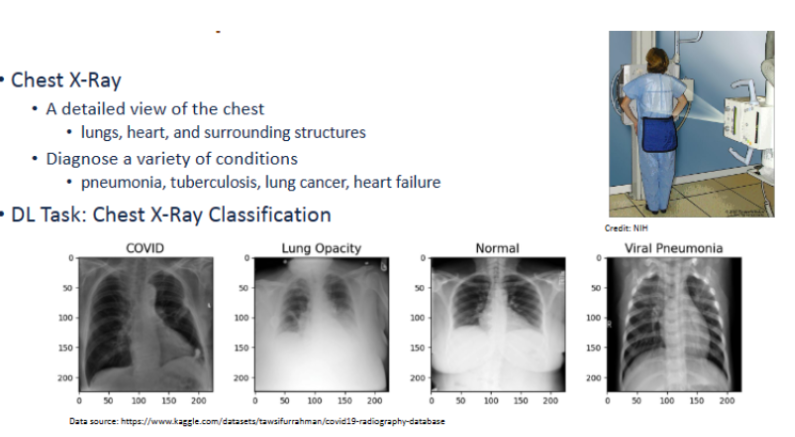
真实世界数据示例
- 胸部X光检查
- 胸部的详细视图
- 肺、心脏和周围结构
- 诊断各种疾病
- 肺炎、肺结核、肺癌、心力衰竭
- 胸部的详细视图
ResNet
title:ResNet简要介绍
- ResNet是深度学习中一种非常流行的神经网络模型,全称为Residual Network(残差网络)。该网络模型的主要特点是通过引入残差块(Residual Block)来解决深层神经网络中的梯度消失和梯度爆炸问题。传统的深层神经网络由于存在多个非线性变换,信息需要一层层地传递,导致在网络较深时梯度消失等问题,难以训练。
- ResNet通过残差块的设计将神经网络的信息流向相加起来,使得在多个非线性变换的结构中,信息可以直接在某些层之间跳跃,避免了信息在网络层数增加时的损失,达到了更好的效果。
- 另外,ResNet模型的表现也非常优秀,被广泛用于图像识别,目标检测,人脸识别等领域。
临床文本的自然语言处理
- 支持的数据集:MIMIC-III 临床记录、MIMIC-CXR 和 IU-XRay。
- 支持的任务:临床记录分类和医疗报告分类
- 演示了如何从X射线图像生成放射学报告演示代码
医学知识图谱
Medcode(医疗代码)
- 在PyHealth中查找/映射医学概念的工具
- 在PyHealth中提供了疾病分类代码(ICD9、ICD10、CCSCM)、药物分类代码(NDC、RxNorm、ATC),也提供了药物间的相互作用率(DDI),DDI来自论文GAMENet,并不是ATC中DDI的子集
- 方法:
- 在PyHealth中有两个主类:InnerMap和CrossMap,相互独立
- InnerMap:在某一个编码系统内匹配,医学概念的查找
举例:
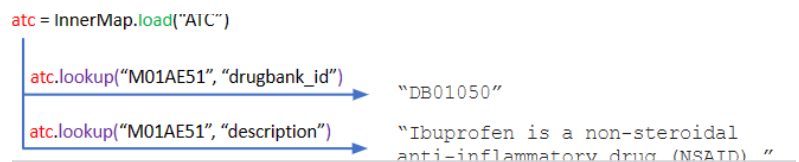
1.加载"ATC"药物编码系统,用lookup()找’M01AE51’对应的药物编码DB01050,找’M01AE51’对应的药物描述:布洛芬是一种抗炎药
2.知识图的感觉
- CrossMap:医疗代码的交叉映射,加载源词汇表和目标词汇表
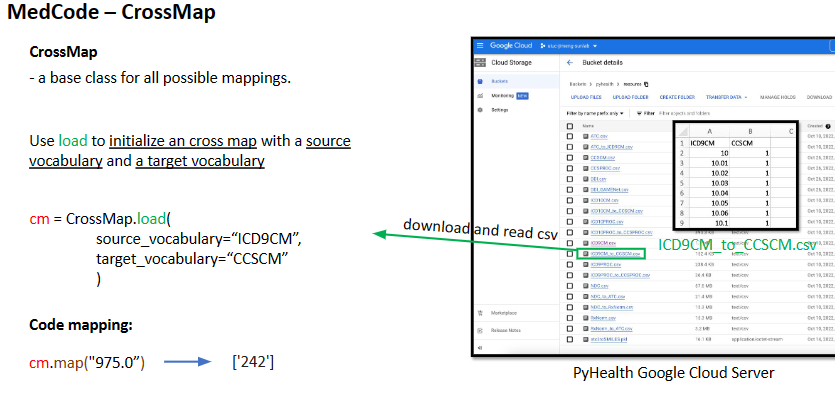
示例中举的是ICD9中编码ID’975’在CCSCM中对应的编码ID’242’
Pre-trained Embedding(预训练嵌入)
将医学概念的语义引入训练
知识图谱嵌入
1.将实体和关系映射到向量空间
输入:知识图谱的三元组(e1,r,e2)
输出:一张完整的知识图,作者称:KGE(知识图的嵌入)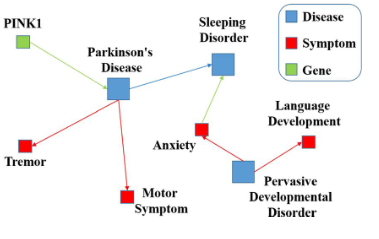
1.输入疾病(帕金森)及症状、基因
好处:
2.在知识图中的实体和关系,编码彼此接近的相似医学概念引入
3.将EHR数据集之外的外部知识被KGE,即使某个数据即的信息并不全面,但是因为引入了KGE,所以对模型预测能力的提升有帮助
语言模型嵌入
文本数据经过语言模型的预先训练,变成预训练词嵌入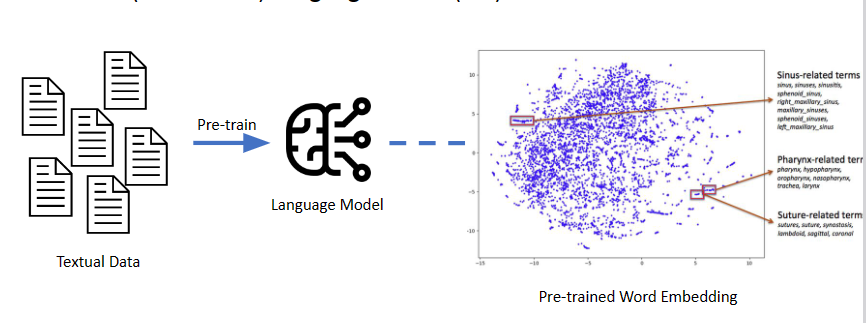
- 支持模型ClinicalBert、BioGPT、SapBert
使用预先训练的嵌入进行基于EHR的预测
做知识图嵌入和语言模型嵌入都是为了增强基于EHR数据模型的预测能力
1.相似的医学概念在向量空间中有相似的嵌入
2.不同医学概念之间的关系被预先建模
在没有预训练嵌入的时候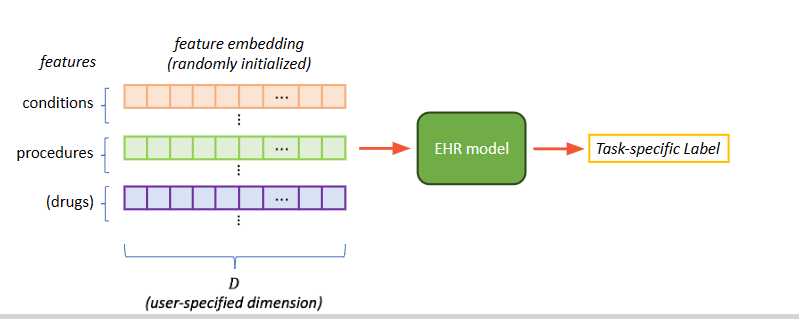
带预训练嵌入的时候: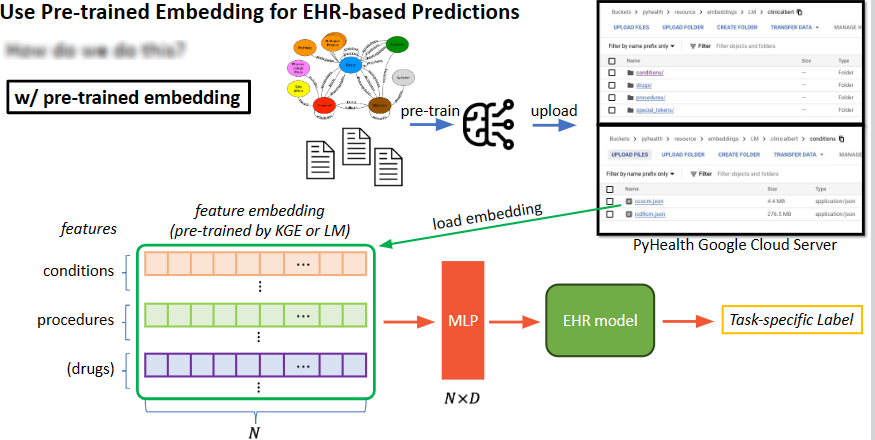
- 都提供了函数去支持他提到的功能
- 演示了利用UMLS 知识图嵌入改进 MIMIC-III 数据集上的药物推荐任务演示代码
使用语言模型生成综合电子病历
- 作者说提供这个功能是因为有一些数据不共享,想要生成一些数据去代替真实的数据,但是他举例的是开源数据库,既然访问真实数据库不方便,那有这个东西没有数据也没有用吧,并且举例的代码中,虽然使用了HALO这个模型,但是好像就是把原始数据库标准化的过程,有点像将MIMIC数据转到P库的样子
代码 - 1.加载了eICU数据集,包括ICD9诊断代码、实验室、用药、治疗、体检、呼吸处理等数据
- 2.只使用了数据集中的诊断和实验室数据集
- 3.在原始数据集中,这两个信息的表存储形式是csv形式,想做的就是利用PyHealth将原始表形式统一转化为python object(他转化成了pyHealth中的数据形式,不过可以通过函数转化为df)
- 4.还是任务流程的5个步骤,其中在选择模型的时候加载的是预先训练好的HALO(分层自回归语言模型合成极高维的纵向电子健康记录),
- 5.结果:

- 通过函数转化为df
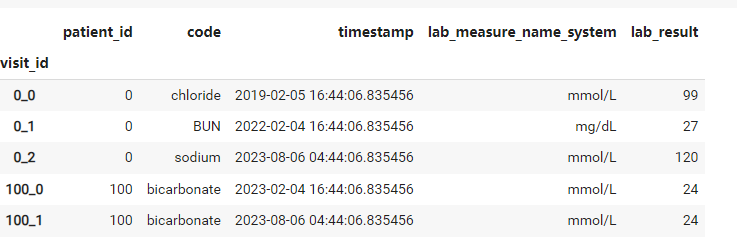
事后不确定性量化
- 常见的事后任务,比如模型校准、预测集构建和预测区间构建
- 校准:模型训练后,为缓解神经网络过拟合或欠拟合的问题
- 在PyHealth中实现的校准方法:温度缩放(过拟合)、直方图分箱、狄利克雷校准、基于内核的校准
演示:code
-
演示了使用温度缩放、直方图分箱和基于内核的校准来校准经过训练的SPaRCNet,用于ISRUC数据集上的睡眠分期任务
-
还演示了使用LABEL(保形预测?)构建预测集,并保证错误覆盖风险,同样是在使用ISRUC数据的睡眠染色任务的SPaRCNet模型上
-
支持将个人数据上传,YouTube教程,可借用插件youtube双语字幕和youtube中文配音观看youtube视频
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容






所有评论(0)